L’équipe de Laszlo Tora en collaboration avec celle d’Evi Soutoglou de l’Institut de génétique et de biologie moléculaire et cellulaire (IGBMC) est parvenue à comprendre un des mécanismes impliqué dans la réparation de l’ADN. Cette découverte a fait l’objet d’une publication dans la revue Journal of Cell Biology.
20/09/2018
Lorsque l’ADN d’une cellule est victime d’une cassure, elle doit le réparer. Soit la réparation s'effectue de manière optimale et la cellule reprend son cycle normal de division, soit la cassure n'est pas réparée correctement et elle provoque l'apparition d'anomalies chromosomiques pouvant entraîner de nombreuses maladies comme le cancer.
Pour réparer les dommages causés à l’ADN, la cellule dispose de plusieurs mécanismes. L’un deux se nomme la recombinaison homologue. Cette réparation nécessite un remodelage des chromosomes, processus dans lequel sont impliqués des complexes déposant ou enlevant des modifications sur des histones, les principaux composants protéiques de la chromatine.
Deux complexes impliqués dans l’expression des gènes
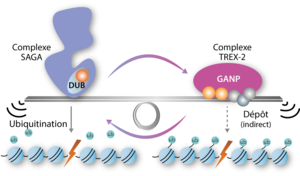
Dans ce mécanisme, les chercheurs ont découvert que l’équilibre induit par les actions antagonistes de deux complexes protéiques impliqués dans l’expression des gènes, nommés TREX-2 et SAGA, était nécessaire pour une réparation efficace de l’ADN. L’élimination de la fonction de TREX-2, diminue la capacité de réparation de l’ADN par recombinaison homologue.
TREX-2 et SAGA ont une sous-unité en commun, appelée ENY2. Lorsque les chercheurs diminuent l’expression de ENY2 l’efficacité de réparation de l’ADN par recombinaison homologue augmente. Une observation qui suggère que le complexe de transcription SAGA est probablement à l’origine de l’augmentation de l’efficacité de la réparation.
Vers une régulation de la réparation de l’ADN dans les cellules cancéreuses
« C’est une sorte de balance qui nécessite les actions antagonistes des deux complexes de transcription pour permettre une recombinaison homologue normale », détaille Laszlo Tora qui, pour parvenir à ce résultat, a réalisé des tests de biologie cellulaire. « Nous avons induit des cassures à l’ADN et avons regardé comment les cellules sont capables de réparer leur ADN en présence ou en l’absence de TREX-2 ou de SAGA. Le tout, sur des cellules humaines primaires ou cancéreuses. »
Cette découverte n’a pas d’applications directes mais va permettre d’ouvrir la voie vers une meilleure compréhension de la régulation de la réparation des cassures ADN dans les cellules normales ou cancéreuses. Prochaine étape : étudier et tenter de comprendre les modifications qui s’opèrent sur les histones au niveau des casseurs d’ADN « A quoi servent-elles, et comment ont-elles été déposées là ? », conclut le chercheur.
Marion Riegert